- Home
- Fabricantes
- University of Washington
- PolyPhred
PolyPhred
O PolyPhred compara sequências baseadas em fluorescência em vestígios de indivíduos diferentes obtidos para identificar locais de heterozigotos para substituições de nucleotídeo único. Suas funções são integradas com Phred, Phrap, e Consed-Autofinish.
Fabricante: University of Washington
Descrição detalhada do produto
SOLICITE SEU ORÇAMENTO
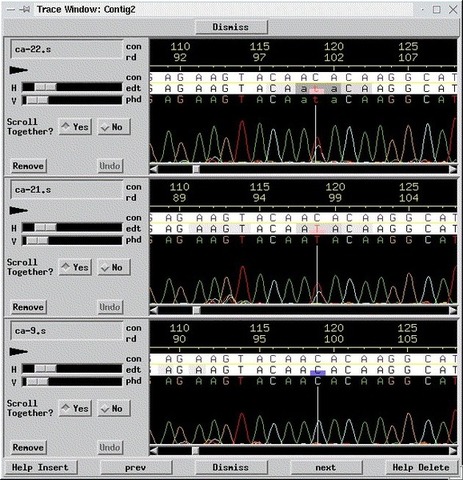
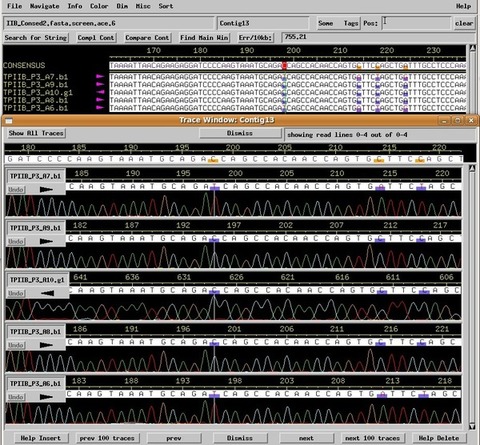
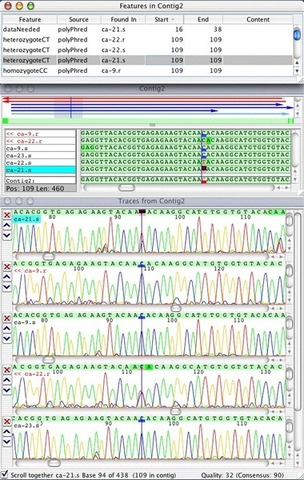
O PolyPhred identifica potenciais locais de heterozigotos, comparando os vestígios em uma montagem de sequência. Phred oferece chamadas-base, informações de qualidade de base e informações de tamanho de pico, que é armazenado em dois tipos de arquivos chamados PHD e arquivos Poly. O Phrap é usado para montar as sequências de entrada para uma ou mais sequências contíguas, e para obter uma sequência de consenso para cada contiguo. As informações são armazenadas em um arquivo chamado arquivo de ACE. PolyPhred usa todos os três tipos de arquivos para analisar os traços de sequência. Ele primeiro lê o arquivo de ACE para obter a sequência de consenso e os nomes dos arquivos de rastreamento (Chromat) utilizados na montagem. Em seguida, ele lê os arquivos PHD e POLY associados a cada traço.
Durante a fase de pesquisa SNP, o software combina informações de todos os traços de sequência traça derivar um genótipo e uma pontuação para cada sequência. Ele também utiliza uma sequência padrão de comparação para identificar os locais que são homozigóticos para um alelo menor ou alternativo. A pontuação indica quão bem o traço no local corresponde ao padrão esperado para um SNP. Depois, o PolyPhred identifica os sítios polimórficos, atualiza os arquivos da ACE e PHD adicionando marcas que destacam as posições dos sites. Os locais marcados podem então ser analisados utilizando o programa Consed. PolyPhred também gera uma saída detalhada que lista as posições, genótipos e pontuações dos sítios polimórficos em um formato que pode ser facilmente analisado em um programa de base de dados.
Parte do processo de computação de pontos envolve uma média de certos valores em toda a leitura na montagem. Para as pequenas montagens, a precisão dessas médias aumenta com o número de sequências que se sobrepõem. Isto por sua vez aumenta a fiabilidade dos resultados. Recomendamos que a região de interesse deva ser abrangida por, pelo menos, oito sequências independentes, se possível.
Um aumento significativo na taxa de SNPs verdadeiros-positivos pode ser conseguidos por sequenciação de cada uma das amostras em ambas as direções. O PolyPhred combina informações de cadeia dupla para aumentar a precisão de suas chamadas de genótipos. Para aproveitar esse recurso, é necessário usar uma convenção de nomenclatura sensível ao nomear os arquivos de dados de sequência. Estes devem conter um conjunto contíguo de caracteres que identificam a fonte individual. Usando a bandeira -fonte, PolyPhred pode então combinar sequências que são da mesma fonte.
Produtos Relacionados
Obrigado! Logo entraremos em contato!